和义广业【行业分析】之测序技术系列,将系统介绍第三代测序技术(单分子测序)各大厂的详细情况。本篇系统介绍国外几家测序公司,其各有特色的测序原理、测序读长、精度,以及公司代表的产品等。
作为当代最热门的测序技术,单分子测序仪的开发在国内外都如火如荼的进行着,下面我们将对国外的几家单分子测序的代表企业及其技术路线进行详细介绍。
(一)Helicos公司
1、公司介绍
作为单分子测序技术的开山之作,由斯蒂芬·奎克(Stephen Quake)教授创办的Helicos公司在2008年推出的Heliscope单分子测序仪由于读长太短、售价太高(测序仪100万美金,试剂48000美元/人),最终在2012年底因为没有获得融资而不得不破产。2013年美国破产法院批准Helicos的请求将知识产权售予Illumina, Life Tech, Fluidigm, Complete Genomics,Helicos公司便从此从人们的视野中消失。
Helicos公司的继任者是瀚海基因,2012年,在奎克实验室完成博士后工作的贺建奎教授回国创立了深圳市瀚海基因生物科技有限公司,继续开展单分子基因测序技术的研发工作,而Helicos的创始人斯蒂芬·奎克教授正是瀚海基因的首席科学顾问。2015年瀚海基因对外公布其单分子基因测序仪GenoCare的原理样机,2017年8月GenoCare正式投产。
2019年8月,深圳市瀚海基因生物科技有限公司官网宣布,公司已正式更名为深圳市真迈生物科技有限公司GeneMind Biosciences Company Limited,简称为“真迈,GeneMind”。后续将对GeneMind进行详细介绍。
2、Helicos单分子测序技术
Helicos遗传分析系统是首个基于荧光测序原理的商业化单分子测序平台,其利用Helicos单分子测序(true single molecule sequencing,tSMS)技术直接测定DNA,能够对几个到几千个碱基的核酸进行测序,但单位质量序列的产量取决于3'端羟基的数量,因此相对较短的模板测序效率较高,对于超过1000nt的核酸,Helicos一般建议将核酸剪切至平均长度为100~200nt[1]。
(1)测序过程[2]
-用于测序的DNA大片段首先被断裂为成千上万的DNA短片段,并在其3'末端添加poly(dA)尾;
-带有poly(dA)尾的DNA链与一次性玻璃flow cell上的oligo(dt)50进行原位杂交,并通过oligo(dt)50带有的荧光信号精确定位;
-每一张标准的flow cell上有25条通道,一旦flow cell装载了适当的样品,它将与合成和成像测序所需的所有试剂一起插入Heliscope测序系统;
-flow cell插入后,拍摄第一张模板图像,通过 oligo(dt)50末端的荧光标记精确定位杂交模板所处的位置;
-逐一加入荧光标记的单色末端终止子和聚合酶孵育,清洗未结合的单色末端终止子,拍摄图像确定每个DNA序列中的一个核苷酸;
-切割荧光染料和抑制基团,切割后,分离的荧光染料被洗掉,然后加入新的聚合酶和一个单荧光核苷酸,拍摄另一张图像,重复这个过程,直到片段完成测序,如图2所示。
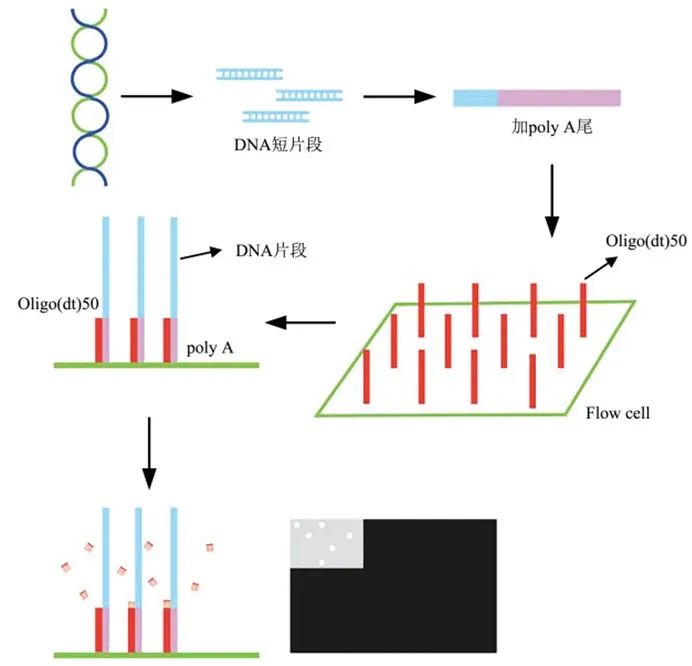
图. Heliscope测序
(2)优势及缺陷分析
-不需要进行PCR扩增,可避免了PCR扩增产生的偏倚和误差;
-对GC含量的敏感性更低,对具有极端GC含量的DNA或基因组测序效率更高;
-所需的试剂和操作步骤较少,只需简单的片段化 DNA、加poly A尾再进行杂交、测序。
-HeliScope读长较短(55-70个碱基);
-数据输出低(20 Gb),尚不及第二代测序仪HiSeq 2000和SOLiD 4;
-测序的错误率较高,虽然可通过重复测序降低错误率,但会造成成本的增加。
(二)Oxford Nanopore
1、公司介绍
Oxford Nanopore成立于2005年,是基于纳米孔单分子实时电信号测序技术的一家公司。于2021年底在伦交所上市,发行价为425便士,募资3.5亿英镑(约4.7亿美元),以发行价计算,Oxford Nanopore市值34亿英镑(约46亿美元)[3]。
2、公司/理念发展
- 公司的测序理念早于1989年由美国加州大学戴维斯分校的David Deamer教授所提出,他猜想:蛋白质通道可能嵌入脂质膜中,而且所得的通道可能容纳核苷酸个体(DNA的小型组份),每个核苷酸在通过通道时都可能会产生特定的离子电流阻断,这一概念后来被Hagan Bayley教授所证实;
- 1991-1996年Dan Branton教授开始对纳米孔传感概念进行研究,发现通过在脂质双层中置入纳米孔可引起核酸移位,并指出“因此通道阻断可用于测量多核苷酸的长度,理论上可以对DNA或RNA单分子中的碱基序列进行直接、高速的检测;
- 2001年,牛津大学Hagan Bayley教授的实验室在《自然·纳米技术》期刊中描述了一种可行性的纳米孔传感器:单链DNA (ssDNA)分子与拴系DNA链结合,可导致通过纳米孔的离子电流发生变化,基于DNA双链体的生存期,不同于单碱基替换,DNA纳米孔能够区分长度达30个核甘酸的单个DNA链;
- 2005年,Oxford Nanolabs作为Oxford Nanopore的前身由Gordon Sanghera博士、Spike Willcocks博士和Hagan Bayley教授(现任牛津大学化学生物学教授)创立。
早在20世纪90年代Deamer等提出使用纳米孔作为生物传感器[4],传感器可分为固态纳米孔和生物纳米孔两大类,已有文献证明这两种类型的纳米孔都能够在单分子水平上检测生物和化学分子。固态纳米孔可以从多种材料如硼、铝、硅、石墨烯以及混合材料中制备,用于进行DNA测序和蛋白质检测;生物纳米孔是一种跨膜蛋白质通道,可以通过改变特定位点氨基酸残基的分子生物学技术进行基因工程改造[5]。
3、Nanopore测序技术
(1)测序原理
-
-
-
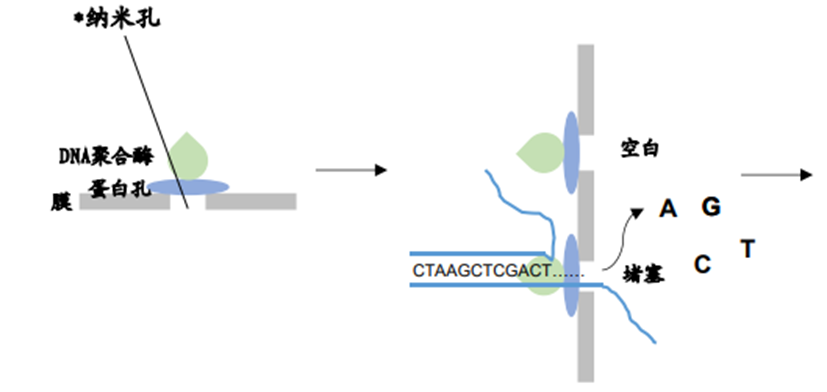
结构:两个电解室通过一层不透水的合成膜分隔开(该膜具有非常高的电阻),将蛋白质纳米孔嵌入合成膜上,用于信号监测。
-
原理:带有蛋白质纳米孔的合成膜浸没在电生理溶液中,当电压施加到电解液室中时,会产生穿过孔的稳态离子电流;进入纳米孔的单分子会对离子的流动造成阻碍(Nanopore信号),不同的碱基造成的阻碍大小是不同的,因此可以检测出不同的碱基。
-
示例:以DNA测序为例,Nanopore测序在文库构建时,在DNA片段上加了一个带有Moter蛋白(DNA解螺旋酶)和Tether蛋白(把DNA链吸附在测序芯片的膜上)的接头;Reader蛋白(构成纳米孔)插在一层电阻率很高的薄膜当中,并浸没在电生理溶液中;当电压施加到电解液室中时,会产生穿过孔的稳态离子电流;当DNA单链通过纳米孔时,会对离子流动造成阻碍,不同碱基造成的阻碍不同,通过分析电流信号可获得DNA序列(如图[7])。
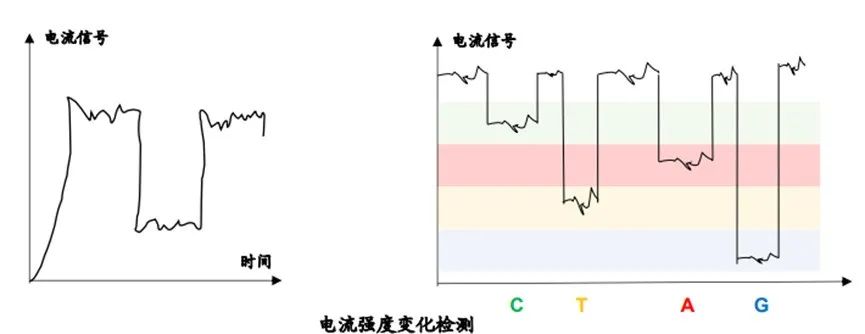
(2)优势及缺陷分析
-测序读长很长,能直接测定1Mb以上的读长(有效地解决二代宏基因组测序技术在病原学诊断领域的缺陷)[9];
-可对单个DNA进行测序,而无需对样品进行PCR扩增或化学标记;
-对于RNA测序而言,无需把RNA逆转录成cDNA,节约时间,降低成本;
-测序通量很高(目前最新版Nanopore的RNA直接测序芯片运行一次可获得 100万条全长RNA序列);
-单芯片的通量恰好适用于1-2个标本,可随时开机测序。
-当一段DNA序列中有较少几个连续相同的碱基(如2个A)时,纳米孔测序识别A 碱基的数量可能会产生误差(测序结果可能为1个A);
-误差率较大(在15%~40%之间),测序错误主要由插入和删除碱基引起。
4、公司代表产品
Oxford Nanopore推出的纳米孔测序装置包括以下4种:MinION、GridION、PromethION、Flongle。
(1)MinION—实时、长读长
MinION是一款便携式、实时、长读长、低成本设备,可用于包括疾病/病原体监测、环境监测、食物链监测、自我量化,甚至微重力生物学等领域的生物分析。

(2)GridION—台式设备
GridION是一款配有计算模块的台式测序仪,具有可运行多达五个MinION测序芯片的能力。

(3)PromethION—大规模、长读长、直接DNA和RNA测序
PromethION是一款模块化的、按需测序设备,测序能力高于MinION的近300倍,可运行高达48个测序芯片

(4)Flongle—MinION和GridION的适配器
Flongle可在较小的一次性测序芯片上进行直接、实时的DNA或RNA测序。

5、其他信息
- 技术关键:其一是合成最小内径只允许DNA单链通过的蛋白纳米孔,且核心区域可容纳通过的碱基数量越少越好(ONT的CsgG是4-6个健基,Illumina的MsgA是3个健基);其二是算法。两者决定了该方法的测序准确度。
- 价格:1000-4000美元
- 准确度:ONT(Oxford Nanopore Technologies)官方数据,现在DNA主要采用1D建库,使用R9.4.1芯片,High-accuracy basecalling(HAC)模式进行DNA测序,原始错误率5%;RNA测序错误率6.1%。HAC是目前最常用的测序模式,市面上的测序公司主要使用这种方式进行测序。双链建库测序测序准确度是96%,但是会减少数据产量,成本升高。
(三)Pacific Biosciences公司
1、公司的发展与合作
- 2003年1月,Science杂志发表了名为 “Zero-Mode Waveguides for Single-Molecule Analysis at High Concentrations” 的文章,ZMW这一概念被提了出来;
- 2009年,时隔6年,Pacific Biosciences的创始人Stephen W. Turner等科学家在Science杂志上发表了题为 “Real-Time DNA Sequencing from Single Polymerase Molecules” 的文章,将ZMW技术应用到了单分子实时测序中;
- 2017年7月,诺禾致源及Pacific Biosciences公司宣布,双方已经就共同开发并推广基于PacBio Sequel测序平台的新型应用程序达成协议;
- 2018年11月1日,Illumina试图以12亿美元收购PacBio,然而,可能由于在2020年1月进行冗长的监管审批程序,该交易被终止[10];
- 2021年,Pacific Bioscience以8亿美元收购高精度测序商Omniome,这是一家开发高度差异化的专有短读长测序平台,其独有的Sequencing by Binding (SBB)测序方法大大提高了测序技术的准确度[11];
- 2021年10月,PacBio与贝瑞基因联合开发的三代测序平台 Sequel II CNDx通过技术要求检验;
- 2022年,贝瑞基因与PacBio签署协议,双方将联合开发全球范围内首款基于第三代测序技术的桌面测序仪,据说这款桌面测序仪小巧便捷,可灵活部署,降低了使用门槛,平台基于SMRT测序技术,能够提供高精度,超长读取,均匀覆盖以及直接检测碱基修饰的技术能力,可精准检测二代基因测序技术(NGS)检测范围外的复杂基因突变类型。此外,整套设备摆放面积较普通测序仪大大减少,成本低且操作简便,具有出色的性价比[12];
- PacBio最近扩大了与Invitae的合作,包括评估SBB平台在肿瘤领域的应用;与Twist Bioscience的合作将开发用于HiFi测序的靶向富集产品;与Broad研究所合作正在开发单细胞RNA isoform测序方法,以增加每次读取的数量,从大约160万到3300万;已经与加拿大Canexia Health公司合作[13]。
2、PacBio单分子测序技术
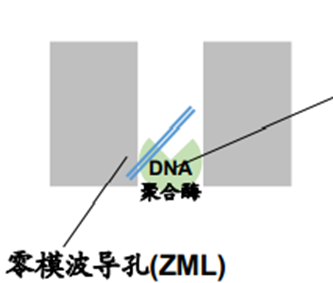
PacBio单分子测序是基于零模波导(zero-mode waveguide,ZMW)特性的单分子实时测序(single-molecule real-time sequencing,SMRT)。ZMW是一种纳米光子封闭结构,由放置在透明二氧化硅基底上的铝包层薄膜中的一个直径约70nm,深度约100nm圆孔组成,ZMW孔的直径小于光的波长(如下图),所以当光通过ZMW孔时,光场呈指数衰减,在被照射的ZMW孔内,产生一个足够小的发光观察体积,包含一个单核苷酸的DNA聚合酶的活性很容易检测出来[14]。
PacBio有两种测序模式:高精度长读(highly accurate long reads,HiFi reads)和连续长读(continuous long read,CLR),见下表。
(1)PacBio单分子测序过程
SMRT是一种并行的单分子DNA 测序方法,以SMRT芯片为载体边合成边测序,其测序过程分为以下几步(如图所示):
-制备SMRTbell模板文库,一种封闭的单链环状DNA,两端加有接头;
-加载到SMRT cell中的SMRTbell扩散到ZMW的测序单元中,单个聚合酶锚定在每个ZMW的底部并与DNA模板、测序引物结合;
-4种不同荧光标记的dNTP底物随机进入ZMW底部,当一种与正要合成的碱基一致的dNTP被DNA聚合酶结合后,激发光从ZMW底部照射进来,使该dNTP发出荧光, 检测器检测核苷酸掺入的荧光信号;
-聚合反应完成后,dNTP上的磷酸基团和荧光基团被切割下来,切割下来的荧光信号扩散出检测体积,不再被检测。
(2)PacBio单分子测序的优势及缺陷分析
-无需进行PCR扩增即可对单个DNA分子进行实时测序;
-长读长,测序数据中有1/2的数据读长大于20 kb,最长读长大于60kb
-测序准确率高,并且无系统误差,即使是高GC含量的区域,测序的偏倚也较少;
-可对AT或GC富集区域以及大的结构变异,包括插入、缺失、倒位、易位、重复和串联重复等难以测序的区域进行测序。
-测序文库构建繁琐,不能直接测定RNA,需要逆转录成cDNA才能进行测序。
3、其他信息
- 关键创新:零模波导孔(zero-mode waveguides, ZMWs)和荧光标记在核苷酸焦磷酸链上(Phospholinked nucleotides)
- 准确度:从天然DNA或RNA分子中捕获序列数据可实现具有99.9%以上的单分子精度的高精度长读
- 近况:公司最近在reads长度和准确性方面取得了突破性进展,数据显示,PacBio可以获得高达200 bp的reads长度且精度高于Q40,大多数reads接近Q50,也就是每10万个碱基中出现一个错误;SBB技术错配错误率非常低,可检测样本和参考基因组之间真正的变异水平 【注:Q20指错误率1%(正确率99%);Q30指错误率0.1%(正确率99.9%);Q40指错误率0.01%(正确率99.99%)】
- 营收:PacBio公布的2021年初步收入为1.305亿美元,同比增长65%[15]
参考文献:
[1] Thompson J F, Steinmann K E. Single molecule sequencing with a HeliScope genetic analysis system. Curr Protoc Mol Biol, 2010, 92(1): 7.10.1-7.10.14
[2] Thompson J F, Steinmann K E. Single molecule sequencing with a HeliScope genetic analysis system. Curr Protoc Mol Biol, 2010, 92(1): 7.10.1-7.10.14
[3] 信息来源:https://baijiahao.baidu.com/s?id=1712315457131273709&wfr=spider&for=pc
[4] Deamer D, Akeson M, Branton D. Three decades of nanopore sequencing.Nat Biotechnol, 2016, 34(5): 518-524
[5] MagiA, Semeraro R, MingrinoA, et al. Nanopore sequencing data analysis: state of the art, applications and challenges. Brief Bioinform, 2018, 19(6): 1256-1272
[6] 视频来源:Oxford Nanopore官网
[7] 俞晓玲, 姜文倩, 郑玲,等. 单分子测序技术及应用研究进展.
[9] Van Dijk E L, Jaszczyszyn Y, Naquin D, et al. The third revolution in sequencing technology.Trends Genet, 2018, 34(9): 666-681
[10] 信息来源:http://news.sohu.com/a/560176885_121359185
[11] 信息来源:仪器信息网 https://www.instrument.com.cn/news/20210720/586067.shtml
[12] 信息来源:https://baijiahao.baidu.com/s?id=1721742794781682321&wfr=spider&for=pc
[13] 信息来源:腾讯网https://new.qq.com/omn/20220116/20220116A02TR400.html
[14] Korlach J, Marks P J, Cicero R L, et al. Selective aluminum passivation for targeted immobilization of single DNA polymerase molecules in zero-mode waveguide nanostructures. Proc Natl Acad Sci USA, 2008, 105(4): 1176-1181
[15] 信息来源:https://new.qq.com/omn/20220116/20220116A02TR400.html
封面图来源:图片来自网络,侵删
作者声明:感谢本文参考资料作者,文中观点仅供参考,不恰当之处还望包涵指正,资料内容侵删。
作者:李芳 武瑾嵘
审核:李芳